“Besitz” und “Vererbung” von “Attributen” in RDFS und OWL#
Über diesen Text: Es gab eine persönliche Kommunikation mit Govdata-Experten aus fitko.de zur Semantik und Pragmatik von Formulierungen und Modellen in der Spezifikation https://www.dcat-ap.de/def/dcatde/2.0/spec, insbesondere
dort zum UML-Diagramm, und
dort zu der Formulierung
Dadurch, dass dcat:Catalog zudem eine Sub-Klasse von dcat:Dataset ist, vererbt dcat:Resource seine Eigenschaften auch auf den dcat:Catalog (Hervorhebung JB, Quelle: Überschrift 4.1 Klasse: Ressource)
Ich stelle hier zur Diskussion ein mögliches Problem mit dem UML-Diagramm und der zitierten Formulierung vor. Es folgt konstruktiv ein Lösungsansatz, wie das Diagramm und die kritische Formulierung auch im Kontext von RDFS und OWL sinnvoll interpretiert werden könnten.
JB 2024-09-18
Abstract#
In RDFS und OWL sind Begriffe wie der “Besitz” von “Eigenschaften” (oder im folgenden “Attributen”) und ihre “Vererbung” nicht definiert. Dennoch wird auf nicht wenigen Websites die Vererbung von Attributen in RDFS oder OWL erklärt – leider unsachgemäß, insbesondere dann, wenn dabei Denkmuster in der Tradition der Objektorientierten Modellierung zur Verwendung kommen. Bisweilen kann man sogar eine Fehlkonzeption von RDFS oder OWL vermuten. (Es ist in der Tat nicht hilfreich, dass in OWL von owl:Class die Rede ist, hier aber eben keine OO-Klassen, sondern Klassen aus der Theorie der Mengenlehre, vereinfacht gesagt als nur Mengen gemeint sind.)
Diskussionen dazu im Netz:
OWL Class and Subclass Property Inheritance https://stackoverflow.com/questions/37854557/owl-class-and-subclass-property-inheritance
Inheritance terminology not appropriate for RDFS and OWL #1228 w3c/dxwg#1228
RDFS/ OWL Inheritance with Joseki+Pellet https://web.archive.org/web/20130831164956/http://answers.semanticweb.com/questions/619/rdfs-owl-inheritance-with-josekipellet
Wir wollen nicht dabei stehen bleiben, in solchen Fällen mahnend den Zeigefinger zu erheben. Denn es scheint ja ein Bedürfnis zu geben, auch im Kontext von RDFS und OWL bestimmte OO-Denkmuster anzuwenden. Also wollen wir versuchen, ein für OO ganz zentrales Denkmuster – nämlich ein Klassendiagramm in UML-Notation – so auf RDFS oder OWL abzubilden, dass eine bestimmte Stärke dieser Modellierung auch in RDFS und OWL praxistauglich erhalten bleibt.
Ein typischer Denkfehler in Bezug auf RDFS oder OWL besteht in der Annahme, dass diese Sprachen geeignet sind, die Integrität von Daten überprüfen zu können. In RDFS ist dies überhaupt nicht möglich, in OWL nur sehr bedingt, jedenfalls nicht praxistauglich. Die Semantic Web Sprache SHACL dient explizit der Integritätsprüfung, erfordert allerdings einiges an Expertenwissen und ist zum derzeitigen Stand der Technik ebenfalls nur bedingt praxistauglich. Es dürfte diese mangelhafte Fähigkeit der Integritätsprüfung mithilfe von RDFS oder OWL sein, die den Einsatz insbesondere von UML-Klassendiagrammen motiviert.
Man muss in einer Pluralität von Expressionen zum selben Denkinhalt nicht unbedingt ein Problem sehen. Im Gegentei: In der Wissenschaft ist durchaus üblich, komplexe Phänomene in verschiedenen Modellierungs-Sprachen zu beschreiben, besonders dann, wenn jede Sprache eine spezifische Erklärungskraft besitzt. Dieses Phänomen wird z.B. ind [] näher beschrieben.
Statt bei einer Pluralität stehen zu bleiben kann man allerdings durchaus versuchen, Modelle aufeinander abzubilden. In unserem Fall wollen wir eine Methode suchen, mit der die in einem UML Klassendiagramm enthaltene Idee des “Besitzes” und der “Vererbung” von “Attributen” sinngemäß in die RDFS oder OWL-Welt übertragen kann. Wir nehmen dabei an, dass der “Sinn” eines UML-Diagramms in unserem Kontext in der Integritätsüberprüfung eines Datensatzes in Bezug auf Pflicht- und optionale Attribute und ihre Vererbung besteht.
Wir entwicklen im folgenden und implementieren ein Zusammenspiel …
von einem nicht zu komplexen OWL Modellierungspattern,
einigen auf das Pattern zugeschnittenen einfachen SPARQL-Queries, und
einigen auf die Ergebnisse dieser SPARQL-Queries zugeschnittenen einfachen Mengen-Ausdrücken.
Die vorliegende Seite liegt in einer Manifestation als lauffähiges Python Notebook (*.ipynb) vor.
Situation#
Spezifikationen wie z.B. https://www.dcat-ap.de/def/dcatde/2.0/spec/ erstellen zur symbolischen Notation einer bestimmten geistigen Vorstellung verschiedene Teil-Expressionen, die sich verschiedener Sprachen bedienen. Zur verwendung kommen
vor allem die natürliche Sprache wie EN und DE
dann auch Sprachen des Semantic Web: “DCAT-AP und damit die deutsche Ableitung DCAT-AP.de ist ein RDF-Vokabular.” (https://www.dcat-ap.de/def/dcatde/2.0/spec/#bedarfsbeschreibung-metadatenstruktur-fur-offene-verwaltungsdaten)
ergänzend auch ein UML-Diagramm (https://www.dcat-ap.de/def/dcatde/2.0/spec/#uml-diagramm)
Mit Kenntnis von FRBR WEMI kann hierin eine Expression erkennen, die aus verschiedenen Teil-Expressionen besteht,
von denen keine einzige das Gesamtwerk alleine ausdrücken kann,
die sich aber ergänzen und insgesamt zu einer umfassenderen, differenzierteren Darstellung der eigentlichen Idee beitragen.
Eine solche Trias aus natürlicher Sprache, Semantik-Web-Sprachen und UML kommt auch in der übergeordneten Decat-Spezifikation (EN, https://www.w3.org/TR/vocab-dcat-2/) und auch in der ersten Version von 2014 (https://www.w3.org/TR/2014/REC-vocab-dcat-20140116/) zur Verwendung.
RDFS-Ontologien und UML-Klassendiagramme folgen fundamental unterschiedlichen Meta-Datenmodellen. Die Specs verwenden grundlegend unterschiedliche Modellierungssprachen, von denen jede bestimmte Dinge gut ausdrücken kann, andere nicht.
UML ist bestens bekannt in der Welt der objektorientierten Modellierung. Man spricht davon, dass Klassen bestimmte Attribute “haben” oder “besitzen” und diese an Unterklassen “vererben”. Hier liegt ein Frame-basiertes Daten-Metamodell zugrunde. Attribute sind Anhängsel einer sie definierenden Klasse; Attribute ohne eine zugehörige Klasse sind nicht denkbar.
In der Semantik Websprache OWL gibt es das Konzept
owl:class. Der Klassenbegriff hier stammt aus der formalen Logik. Etwas vereinfacht gesagt handelt es sich hierbei tatsächlich einfach um Mengen. Eine Klasse in OWL ist einfach eine Menge und hat mit dem Klassenbegriff der objektenorientierten Programmierung sehr wenig zu tun. In der weniger ausdrucksmächtigen Schemasprache RDFS gibt es überhaupt keine Klassen (im Sinne von Mengen), sondern nur die Propertyrdfs:type. Nicht in RDFS und auch nicht in OWL werden Klassen benötigt, um Attribute zu “definieren”. Im RDF-Datenmodell gibt es Properties. Properties sind First Class Citizens, und können auch ohne zugehörige Klassen bestehen.
Wenn man verschiedene Sprachen verwendet, um sich ergänzende Modelle zum selben Gegenstandsbereich zu erstellen, besteht die Gefahr, dass sich die Modelle in bestimmten Aspekten auch widersprechen. inkompatibel sind diese Sprachen insbesondere in Bezug auf den Klassenbegriff. Die Rede vom “Besitz” oder der “Vererbung” von Attributen hat mit RDF und OWL nichts zu tun. Wer naiv aus einem Datenbankhintergrund oder mit einem objektorientierten Verständnis diese Begriffe auf RDFS oder OWL anwendet, begeht einen Fehler.
Tatsächlich wird in https://www.w3.org/TR/vocab-dcat-2/#h-note-1 darauf hingewiesen, dass das UML-Diagramm nur als eine informelle Expression im Sinne von RDF zu verstehen ist:
while the diagram uses UML-style class notation it should be interpreted following the usual RDF open-world assumptions around the presence/absence of properties, relationships, and their cardinality. The properties shown in each class reflect those specified in the descriptions of classes in § 6. Vocabulary specification. To assist in understanding the full scope of each class, properties are copied down from each ‘::super-class’. Cardinalities are shown in a few places to reinforce expectations, but these are not axiomatized or enforced in any way by this (normative) recommendation.
Der “Besitz” von “Attributen” und ihre “Vererbung” sind in RDFS oder OWL nicht definiert. Die Herausforderung besteht also darin, diese aus der objektorientierten Modellierung stammenden Begriffe im abstrakten Daten-Metamodell der Sprachen des Semantic Web sinnvoll zu interpretieren und zu modellieren. Idealerweise gelingt es uns, diese Interpretationen in Form eines lauffähigen funktionalen Prototyps zu operationalisieren. Wir wollen das an dieser Stelle nicht abstrakt und allgemein tun, sondern konkret und spezifisch im Kontext der Aufgaben und Anliegen einer Spezifikation wie DCAT.
Beispiel#
Manchmal wird im Kontext von RDF von Vererbung gesprochen. Gemeint ist damit:
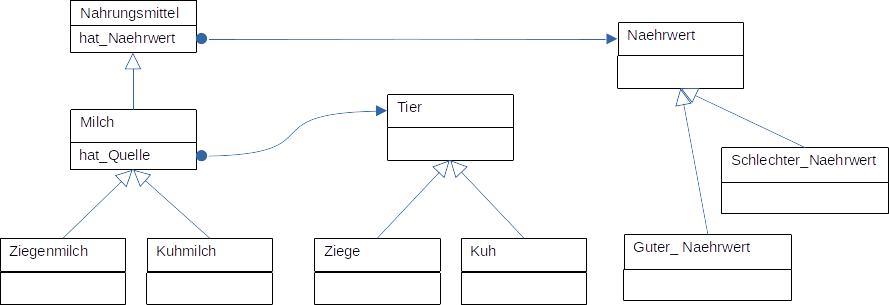
Wenn eine Klasse (zum Beispiel
:Nahrungsmittel) ein bestimmtes Attribut (zum Beispiel:Nährwert) “besitzt”dann “vererbt” es dieses Attribut auch an seine Subklassen (z.B.
:Milch).
Zwei Fragen: Was ist mit “besitzt” gemeint, was mit “vererben”? konkret in unserem Beispiel: Was ist gemeint, wenn man sagt, eine Klasse wie Nahrungsmittel “besitzt” das Attribut “Nährwert”?
Grundlagen Inferencing rdfs:subClassOf#
Dieser Abschnitt: Wiederholung “Was heißt inferencing?” am Bsp. von ISA, rdfs:subClassOf, Regel rdfs9 mit rdflib und owlrl.
import rdflib
import owlrl
Wir erstellen zunächst eine T-Box (owl: terminology box, also in etwa das Schema). Hierfür reicht plain vanilla RDFS vollkommen aus, denn wir verwenden keine komplexeren Axiome.
#namespaces
ns = """
@prefix : <http://example.org/ns#> .
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix owl: <http://www.w3.org/2002/07/owl#> .
@prefix xsd: <http://www.w3.org/2001/XMLSchema#> .
"""
tbox = ns + """
:Nahrungsmittel a owl:Class .
:Milch a owl:Class ;
rdfs:subClassOf :Nahrungsmittel .
:Ziegenmilch a owl:Class ;
rdfs:subClassOf :Milch .
:Kuhmilch a owl:Class ;
rdfs:subClassOf :Milch .
"""
tbox += """
:Tier a owl:Class .
:Ziege a owl:Class ;
rdfs:subClassOf :Tier .
:Kuh a owl:Class ;
rdfs:subClassOf :Tier .
"""
tbox += """
:Naehrwert a owl:Class .
:Guter_Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert .
:Schlechter_Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert .
"""
g1 = rdflib.Graph().parse(data=tbox)
print(f"g1 has initially {len(g1)} triples")
g1 has initially 17 triples
Wir legen dann ausgewählte Instanzen an. Es entsteht eine Reihe von A-Boxen (owl: abox, assertion box). Um die Lesbarkeit zu erhöhen, haben alle Instanzen den Namen der Klasse, ergänzt durch eine laufende Nummer, also z.B. :Milch_1.
abox_rdfs = ns + """
:Milch_1 a :Milch .
:Ziegenmilch_1 a :Ziegenmilch .
:Ziegenmilch_2 a :Ziegenmilch ;
:hat_Naehrwert :Guter_Naehrwert_2 .
:Guter_Naehrwert_2 a :Guter_Naehrwert .
"""
print(abox_rdfs)
@prefix : <http://example.org/ns#> .
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix owl: <http://www.w3.org/2002/07/owl#> .
@prefix xsd: <http://www.w3.org/2001/XMLSchema#> .
:Milch_1 a :Milch .
:Ziegenmilch_1 a :Ziegenmilch .
:Ziegenmilch_2 a :Ziegenmilch ;
:hat_Naehrwert :Guter_Naehrwert_2 .
:Guter_Naehrwert_2 a :Guter_Naehrwert .
Wir fügen diese A-Box zusätzlich zu unserem Graphen g1 hinzu.
g1.parse(data=abox_rdfs)
print(f"g1 has now {len(g1)} triples")
g1 has now 22 triples
Kontrolle: Wie sieht unser Graph jetzt aus? Wir nutzen die Serialisierung von RDFlib.
print(g1.serialize())
@prefix : <http://example.org/ns#> .
@prefix owl: <http://www.w3.org/2002/07/owl#> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
:Kuh a owl:Class ;
rdfs:subClassOf :Tier .
:Kuhmilch a owl:Class ;
rdfs:subClassOf :Milch .
:Milch_1 a :Milch .
:Schlechter_Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert .
:Ziege a owl:Class ;
rdfs:subClassOf :Tier .
:Ziegenmilch_1 a :Ziegenmilch .
:Ziegenmilch_2 a :Ziegenmilch ;
:hat_Naehrwert :Guter_Naehrwert_2 .
:Guter_Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert .
:Guter_Naehrwert_2 a :Guter_Naehrwert .
:Nahrungsmittel a owl:Class .
:Naehrwert a owl:Class .
:Tier a owl:Class .
:Ziegenmilch a owl:Class ;
rdfs:subClassOf :Milch .
:Milch a owl:Class ;
rdfs:subClassOf :Nahrungsmittel .
Wir stellen fest, dass Ziegenmilch_1 zwar ein Element der Menge Ziegenmilch ist, nicht jedoch der Menge Milch. Weil die Menge Ziegenmilch aber eine Teilmenge der Menge Milch ist, sollte jdes Element aus der Menge Ziegenmilch auch ein Element der Menge Milch sein.
Ganua dieses Inferencing erfolgt durch die Regel rdfs9 in der RDFS-Spezifikation https://www.w3.org/TR/rdf11-mt/#patterns-of-rdfs-entailment-informative:
rdfs9:
If S contains:
xxx rdfs:subClassOf yyy . zzz rdf:type xxx .then S RDFS entails
zzz rdf:type yyy .
Wir wollen diese Information ableiten. Dazu brauchen wir eine Inferencing Engine. Wir verwenden owlrl.
(Um einen Vorher-Nachher-Vergleich zu ermöglichen, legen wir dazu einen neuen Graphen an, in den wir unsere bisherigen Daten identisch einlesen.)
g1_nachher = rdflib.Graph().parse(data=tbox)
g1_nachher.parse(data=abox_rdfs)
print(f"g1_nachher has now {len(g1_nachher)} triples")
owlrl.DeductiveClosure(owlrl.OWLRL_Semantics,
axiomatic_triples = False).expand(g1_nachher)
print(f"After inferencing g1_nachher has {len(g1_nachher)} triples")
g1_nachher has now 22 triples
After inferencing g1_nachher has 195 triples
Wie sieht g1_nachher nach dem Inferencing aus?
Die folgende Zelle würde eine recht umfangreiche Ausgabe generieren. Aus Gründen der Darstellung fügen wir diese Ausgabe am Ende dieser Seite an.
#print(g1_nachher.serialize())
Wir sehen, dass owlrl alle Tripel “ausmaterialisiert”, die überhaupt denkbar sind. Nicht an allen sind wir im folgenden interessiert. Wir definieren uns eine kleine Funktion focus(), die in einer rdflib-Serialisierung alle Absätze ausgibt, in der Substrings vorkommen, die wir in einer Liste von interessanten Substrings angeben können.
def focus(focus_string_list, ttl):
return "\n\n".join( [ paragraph for paragraph in ttl.split("\n\n") \
if any( [ focus_string in paragraph for focus_string in focus_string_list ] ) ] )
Alle Absätze, in denen der Substring “Ziegenmilch” vorkommt:
print( focus( ["Ziegenmilch"], g1_nachher.serialize() ))
:Ziegenmilch_1 a :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
owl:sameAs :Ziegenmilch_1 .
:Ziegenmilch_2 a :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
:hat_Naehrwert :Guter_Naehrwert_2 ;
owl:sameAs :Ziegenmilch_2 .
owl:Nothing a owl:Class ;
rdfs:subClassOf :Guter_Naehrwert,
:Kuh,
:Kuhmilch,
:Milch,
:Naehrwert,
:Nahrungsmittel,
:Schlechter_Naehrwert,
:Tier,
:Ziege,
:Ziegenmilch,
owl:Nothing,
owl:Thing ;
owl:equivalentClass owl:Nothing ;
owl:sameAs owl:Nothing .
:Ziegenmilch a owl:Class ;
rdfs:subClassOf :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
owl:equivalentClass :Ziegenmilch ;
owl:sameAs :Ziegenmilch .
Wir sehen, dass die uns interessierende Instanz :Ziegenmilch_1 nun korrekt auch als :Milch (und konsequenterweise als :Nahrungsmittel und owl:Thing eingeordnet wurde: Unsere Inferencing Engine funktioniert!
Modellierung von “besitzt”#
Wir wollen nun die Idee von “Klasse C besitzt das Attribut A” sinngemäß in OWL modellieren.
Unsere Modellierung definiert dazu in OWL …
die Menge 1
:ATT:hat_Naehrwertals die Menge aller Dinge, die ein Attribut:hat_Naehrwertmit einem beliebigen Wert haben.
In einem zweiten Schritt werden wir über eine SPARQL-Query und einfache Set-Operationen in Python zwei weitere Mengen berechnen, nämlich …
die Menge 2
:Nahrungsmittelgeschnitten mit der Menge:ATT:hat_Naehrwert: Das sind alle Nahrungsmittel, für die tatsächlich ein Mehrwert angegeben ist.die Menge 3 aller
:Nahrungsmittel, die nicht auch in:ATT:hat_Naehrwertenthalten sind (also Nahrungsmittel ohne ATT:hat_Naehrwert): Das sind alle Nahrungsmittel, für die kein Mehrwert angegeben ist.
Schritt 1: Restriction definieren#
Für Experten: Wir verwenden im folgenden das OWL-Modellierungspattern Pattern 2: Values as subclasses partitioning a “feature” in der Repräsentations-Variante 1 Using a fact about the individual, wie es in W3C Working Group Note 17 May 2005: Representing Specified Values in OWL > Pattern 2: Values as subclasses partitioning a ‘feature’ beschrieben ist.
Allerdings verzichten wir in unserer Modellierung auf owl:disjointWith, um ein streng monotones Inferencing ohne Negation gewährleisten zu können. Statt die Inferencing Engine mit dem Test auf Disjointness zu betrauen, fragen wir die Disjoint-Eigenschaft später aus einer Applikation heraus durch eine SPARQL-Query ab und geben bei Bedarf ein Integritätsverletzung bekannt.
Um obiges Pattern zu realisieren, erweitern wir zunächst obige T-Box um eine geeignete Restriction. Wir benennen diese künstliche Menge, indem wir ein :ATT_ den Namen voranstellen, also hier :ATT:hat_Naehrwert
tbox += """
:ATT:hat_Naehrwert
a owl:Restriction ;
owl:onProperty :hat_Naehrwert ;
owl:someValuesFrom :Naehrwert .
:hat_Naehrwert a owl:ObjectProperty .
"""
tbox += """
:ATT:hat_Fettgehalt
a owl:Restriction ;
owl:onProperty :hat_Fettgehalt ;
owl:someValuesFrom xsd:decimal .
:hat_Fettgehalt a owl:DatatypeProperty .
"""
:ATT:hat_Naehrwert ist eine Menge. Sie enthält aller Elemente aller Mengen, für die es einen Wert der Property :hat_Naehrwert gibt, der ein Element aus der Menge :Naehrwert ist.
Für Testzwecke legen wir in einer neuen, für das OWL Beispiel spezifischen A-Box einige Instanzen an:
abox_owl = ns + """
:Naehrwert_2 a :Naehrwert .
:Nahrungsmittel_21 a :Nahrungsmittel .
:Nahrungsmittel_22 a :Nahrungsmittel ;
:hat_Naehrwert :Naehrwert_2 .
:Irgendetwas_23 a :Irgendetwas ;
:hat_Naehrwert :Naehrwert_2 .
:Ziegenmilch_2 a :Ziegenmilch ;
:hat_Naehrwert :Naehrwert_2 .
:Kuhmilch_2 a :Kuhmilch ;
:hat_Fettgehalt "3.6"^^xsd:decimal .
"""
Wir nehmen folgende Unterschiede zur Kenntnis:
Nahrungsmittel_21 wird zwar als ein Nahrungsmittel deklariert, es ist jedoch kein Naehrwert angegeben: diese Instanz “besitzt” kein Attribut Naehrwert.
Anders das Nahrungsmittel_22: Dieses wird als ein Nahrungsmittel deklariert; und es ist der Naehrwert
:Naehrwert_2(ein Element der Menge:Naehrwert) angegeben ist. Diese Instanz “besitzt” das Attribut Naehrwert.Interessant ist die Instanz
:Irgendetwas_23, Element einer sonst nicht weiter bekannnten Menge:Irgendetwas. Wir wissen nichts genaues über diese Instanz, außer dass auch sie einen Nährwert hat, nämlich:Naehrwert_2.Für unsere
:Ziegenmilch_2ist den Naehrwert bekannt, nicht aber für unsere Kuhmilch_2 – auch hier fehlt der Naehrwert.
Wir lesen auch diese T-Box und A-Box in einen neuen Graphen ein:
g2 = rdflib.Graph()
g2.parse(data=tbox)
g2.parse(data=abox_owl)
<Graph identifier=N398076f0be77477282a5ab4849996093 (<class 'rdflib.graph.Graph'>)>
Was wissen wir über :Nahrungsmittel_22?
print( focus( ["Nahrungsmittel_22"], g2.serialize() ))
:Nahrungsmittel_22 a :Nahrungsmittel ;
:hat_Naehrwert :Naehrwert_2 .
EXKURS owl:DatatypeProperty
Funktioniert das alles auch mit Properties, die Literale als Werte haben, mithin also eine owl:DatatypeProperty?
print( focus( ["Kuhmilch_2"], g2.serialize() ))
:Kuhmilch_2 a :Kuhmilch ;
:hat_Fettgehalt 3.6 .
q = """SELECT ?example WHERE {?example a :ATT:hat_Fettgehalt .}"""
result = g2.query(q)
print(f"Anzahl Tripel: {len(result)=}")
for row in result:
print( row[0].n3() )
Anzahl Tripel: len(result)=0
ENDE EXKURS, weiter im Text:
Wir erzeugen wie oben eine identische Kopie, die wir dann wie oben dem Inferencing zuführen:
g2_nachher = rdflib.Graph().parse(data=tbox)
g2_nachher.parse(data=abox_owl)
print(f"g2_nachher has now {len(g2_nachher)} triples")
owlrl.DeductiveClosure(owlrl.OWLRL_Semantics,
axiomatic_triples = False).expand(g2_nachher)
print(f"After inferencing g2_nachher has {len(g2_nachher)} triples")
g2_nachher has now 35 triples
After inferencing g2_nachher has 234 triples
print( focus( ["Nahrungsmittel_22"], g2_nachher.serialize() ))
:Nahrungsmittel_22 a <http://example.org/ns#ATT:hat_Naehrwert>,
:Nahrungsmittel,
owl:Thing ;
:hat_Naehrwert :Naehrwert_2 ;
owl:sameAs :Nahrungsmittel_22 .
Interessant ist die Zeile :Nahrungsmittel_22 a :ATT:hat_Naehrwert: Unsere Restriktion hat funktioniert!
EXKURS owl:DatatypeProperty
q = """SELECT ?example WHERE {?example a :ATT:hat_Fettgehalt .}"""
result = g2_nachher.query(q)
print(f"Anzahl Tripel: {len(result)=}")
for row in result:
print( row[0].n3() )
Anzahl Tripel: len(result)=1
<http://example.org/ns#Kuhmilch_2>
ENDE EXKURS
Schritt 2: Mengenoperationen#
Wir prüfen nach, welche Instanzen insgesamt in der Menge :ATT:hat_Naehrwert enthalten sind. rdflib stellt uns dazu eine SPARQL-Schnittstelle zur Verfügung.
q = """SELECT ?example WHERE {?example a :ATT:hat_Naehrwert .}"""
for row in g2_nachher.query(q):
print( row[0].n3() )
<http://example.org/ns#Nahrungsmittel_22>
<http://example.org/ns#Ziegenmilch_2>
<http://example.org/ns#Irgendetwas_23>
Wir können die Ergebnisse dieser Query auch in einem Python-Set abspeichern:
ATT_hat_Naehrwert = { row[0].n3() for row in g2_nachher.query(q) }
ATT_hat_Naehrwert
{'<http://example.org/ns#Irgendetwas_23>',
'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
Entsprechend speichern wir alle instanzen aus :Nahrungsmittel in einem eigenen Set:
Nahrungsmittel = { row[0].n3()
for row in g2_nachher.query("""SELECT ?example WHERE {?example a :Nahrungsmittel .}""") }
Nahrungsmittel
{'<http://example.org/ns#Kuhmilch_2>',
'<http://example.org/ns#Nahrungsmittel_21>',
'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
Die Schnittmenge zeigt uns zu welchen Nahrungsmitteln ein Nährwert angegeben ist.
Nahrungsmittel__INTERSECT__ATT_hat_Naehrwert = Nahrungsmittel & ATT_hat_Naehrwert
Nahrungsmittel__INTERSECT__ATT_hat_Naehrwert
{'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
Die Differenzmenge zeigt uns, zu welchen Instanzen kein Naehrwert angegeben ist:
Nahrungsmittel__MINUS__ATT_hat_Naehrwert = Nahrungsmittel - ATT_hat_Naehrwert
Nahrungsmittel__MINUS__ATT_hat_Naehrwert
{'<http://example.org/ns#Kuhmilch_2>',
'<http://example.org/ns#Nahrungsmittel_21>'}
Wenn nun verlangt wäre, dass zu jedem Nahrungsmittel – einschließlich allen Unterklassen – der Nährwert angegeben sein muss, dann könnten wir so alle Instanzen identifizieren, die diese Bedingung erfüllen oder verletzen.
Auch eine Verallgemeinerung ist möglich: Wir können uns vorstellen, dass von einer Anwendung bestimmte Attribute übergeben werden, die allerdings nicht in die Datenbank aufgenommen werden dürfen. So hat z.B. in einer Adressdatenbank die sexuelle Orientierung einer Person nichts zu suchen, eine entsprechende ATT-Menge müsste also leer sein.
Diskussion: Statt der Mengen-Operationen hätte man auch spezifischere SPARQL-Queries formulieren können. Beispielsweise generiert die folgende SPARQL-Query die Menge Nahrungsmittel__INTERSECT__ATT:hat_Naehrwert direkt:
q = """SELECT ?example WHERE {?example a :ATT:hat_Naehrwert, :Nahrungsmittel .}"""
for row in g2_nachher.query(q):
print( row[0].n3() )
<http://example.org/ns#Nahrungsmittel_22>
<http://example.org/ns#Ziegenmilch_2>
Es stellt sich die Frage,
welche Logik man direkt in der Ontologie modelliert und zur Bearbeitung an die Inferencing Engine übergibt,
welche Logik man über SPARQL-Queries abdeckt, und
welche Logik man programmatisch mit Mengen-Operatoren über die Query-Resultate abdeckt.
In unserer Beispiel-Implementierung verwenden wir mit owlrl eine Inferencing Engine, die das Profil OWL2-RL abdeckt – also im Prinzip ein vergleichsweises ausdrucksmächtiges Profil. Dennoch gehen wir in unserer Lösung davon aus,
dass wir nur ein sehr ausdrucksschwaches Profil verwenden,
das ungefähr im Bereich OWL1 lite liegt, und
das insbesondere keine Negation, kein Disjoint und ähnliches kennt, sondern
dass wir solche Elemente über SPARQL-Anfragen “von außen” abdecken, aber nicht der Inferencing Engine überlassen.
Übergreifendes Ziel: Schnelles, sicheres, monotones Inferencing.
Vorläufiges Ergebnis:
Eine Klasse “besitzt” ein “Attribut”, wenn in der obigen Modellierung eine Instanz dieser Klasse in der entsprechenden Schnittmenge von Klasse und ATT-Restriktion (in unserem Beispiel die Menge
:Nahrungsmittel__INTERSECT__ATT:hat_Naehrwert) enthalten ist.Ein solche Attribut wird “vererbt”, indem eine entsprechende Inferencing Engine Instanzen einer Teilmenge auch als Instanzen entsprechender Obermengen einordnet. Man kann – wenn man partout von Vererbung reden will – dann sagen, dass auch die Subklasse das entsprechende “Attribut” “erbt” und daher “besitzt”.
Als ein “Attribut” kann man dann eine RDF(S) oder OWL-Property bezeichnen, für die eine entsprechende Modellierung bestehend aus ATT-Restriction, SPARQL Queries und Mengenoperationen angelegt wurde.
Wir können nun leicht die Integrität bezogen auf das Attribut :hat_Naehrwert prüfen:
Wenn für ein Element der Menge Nahrungsmittel notwendig einen Nährwert angegeben sein muss, dann müssen alle Elemente aus der Menge Nahrungsmittel auch in der Menge 2 enthalten sein: Die Schnittmenge muss also gleichmächtig sein wie die Menge der Nahrungsmittel, die Differenzmenge muss leer sein.
Wenn hingegen der Nährwert von Nahrungsmittel nur optional angegeben sein muss, dann darf auch die Differenzmenge Elemente enthalten.
rfc2119:MUST#
Offen ist, wie man z.B. in einem Validator operationalisiert, dass ein Attribut angegeben sein muss oder kann oder auch nicht darf.
Üblicherweise wird die Unterscheidung zwischen Pflicht- und optionalen Attributen über die Kardinalität kommuniziert:
Bei einer Kardinalität von einem oder mehr Elementen muss mindestens ein Wert angegeben sein. Es handelt sich also um ein Pflichtattribut, die Menge 3 muss leer sein.
Bei einer Kardinalität von null oder mehr Elementen muss kein Wert angegeben sein. Es handelt sich also um ein optionales Attribut, die Menge 3 darf Elememte enthalten.
Wir suchen hier einen alternativen Weg. Gedankenexperiment: Wir interpretieren die rfc2119-Begriffe als Modalbegriffe und ziehen sie zur Integritäts-Kontrolle heran.
https://www.rfc-editor.org/rfc/rfc2119.txt:
MUST This word, or the terms “REQUIRED” or “SHALL”, mean that the definition is an absolute requirement of the specification.
MUST NOT This phrase, or the phrase “SHALL NOT”, mean that the definition is an absolute prohibition of the specification.
SHOULD This word, or the adjective “RECOMMENDED”, mean that there may exist valid reasons in particular circumstances to ignore a particular item, but the full implications must be understood and carefully weighed before choosing a different course.
gegeben:
eine Menge, z.B.
Nahrungsmitteleine (hier: Object-) Property, hier
hat_Naehrwertein Syntaxelement, das dieser Property in Bezug auf die angegebene Menge einen rfc2119-Begriff zuordnet.
Eine informelle Syntax könnte etwa so aussehen:
Nahrungsmittel
ATT hat_Naehrwert MODALITY must
Die informelle Semantik von must würde lauten:
Wenn zu einer Instanz der Menge Nahrungsmittel kein Nährwert angegeben ist, markiere diese Instanz als “fehlerhaft”.
Wir interpretieren must und must not also so, dass Verletzungen der Modalität zu einer Fehlermeldung führen müssen, resp. ein Datensatz zurückgewiesen werden muss. Technisch:
must not: Die fehlerhaften Elemente von
:Nahrungsmittelbezogen auf:hat_Naehrwertsind exakt die Elemente der Schnittmenge von:Naehrwertund:ATT:hat_Naehrwert.must: Die fehlerhaften Elemente der Menge
:Nahrungsmittelbezogen auf:hat_Naehrwertsind, wie oben gezeigt, die Elemente der Differenzmenge:Nahrungsmittelminus:ATT:hat_Naehrwert.
Ergänzend könnten wir should als eine (starke) Empfehlung interpretieren. Wenn sie verletzt wird, wird der betreffende Datensatz als auffällig markiert, aber nicht zurückgewiesen. Statt dessen reparieren ihn falls möglich (z.B. indem wir einen Standardwert einsetzen) und genehmigen eine weitere Bearbeitung.
class MonitoredCategory():
def __init__(self, *, rdf_graph, category, attribute, range_includes):
self.graph1 = rdf_graph
# print(self.graph1.serialize())
# make a deep copy of graph1
self.graph2 = rdflib.Graph()
self.graph2.parse( data= self.graph1.serialize() )
att_class = ":ATTRIBUTE_" + attribute
att_class_restriction = ns + f"""{att_class} a owl:Restriction ;
owl:onProperty {attribute} ;
owl:someValuesFrom {range_includes} . """
self.graph2.parse(data=att_class_restriction)
# perform inferencing
owlrl.DeductiveClosure(owlrl.OWLRL_Semantics, axiomatic_triples = False).expand(self.graph2)
self.queries = {}
self.queries["category"] = f"""SELECT ?example WHERE {{?example a {category} .}}"""
self.queries["att_class"] = f"""SELECT ?example WHERE {{?example a {att_class} .}}"""
self.instances = {}
self.instances["category"] = { row[0].n3() for row in self.graph2.query(self.queries["category"]) }
self.instances["att_class"] = { row[0].n3() for row in self.graph2.query(self.queries["att_class"]) }
self.faulty = {}
# must not: instances of `category`, which are also instances of `mustnot`, are faulty:
self.faulty["mustnot"] = self.instances["category"] & self.instances["att_class"]
# must: instances of category, which are not also instances of the att_class, are faulty:
self.faulty["must"] = self.instances["category"] - self.instances["att_class"]
len(g2)
35
mc = MonitoredCategory(rdf_graph=g2, category=":Nahrungsmittel", attribute=":hat_Naehrwert", range_includes=":Naehrwert")
mc.queries
{'category': 'SELECT ?example WHERE {?example a :Nahrungsmittel .}',
'att_class': 'SELECT ?example WHERE {?example a :ATTRIBUTE_:hat_Naehrwert .}'}
mc.instances["category"]
{'<http://example.org/ns#Kuhmilch_2>',
'<http://example.org/ns#Nahrungsmittel_21>',
'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
mc.instances["att_class"]
{'<http://example.org/ns#Irgendetwas_23>',
'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
mc.faulty["must"]
{'<http://example.org/ns#Kuhmilch_2>',
'<http://example.org/ns#Nahrungsmittel_21>'}
mc.faulty["mustnot"]
{'<http://example.org/ns#Nahrungsmittel_22>',
'<http://example.org/ns#Ziegenmilch_2>'}
g1_nachher#
print(g1_nachher.serialize())
@prefix : <http://example.org/ns#> .
@prefix owl: <http://www.w3.org/2002/07/owl#> .
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix xsd: <http://www.w3.org/2001/XMLSchema#> .
:Milch_1 a :Milch,
:Nahrungsmittel,
owl:Thing ;
owl:sameAs :Milch_1 .
:Ziegenmilch_1 a :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
owl:sameAs :Ziegenmilch_1 .
:Ziegenmilch_2 a :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
:hat_Naehrwert :Guter_Naehrwert_2 ;
owl:sameAs :Ziegenmilch_2 .
:hat_Naehrwert owl:sameAs :hat_Naehrwert .
rdf:HTML a rdfs:Datatype ;
owl:sameAs rdf:HTML .
rdf:PlainLiteral a rdfs:Datatype ;
owl:sameAs rdf:PlainLiteral .
rdf:XMLLiteral a rdfs:Datatype ;
owl:sameAs rdf:XMLLiteral .
rdf:langString a rdfs:Datatype ;
owl:sameAs rdf:langString .
rdf:type owl:sameAs rdf:type .
rdfs:Literal a rdfs:Datatype ;
owl:sameAs rdfs:Literal .
rdfs:comment a owl:AnnotationProperty ;
owl:sameAs rdfs:comment .
rdfs:isDefinedBy a owl:AnnotationProperty ;
owl:sameAs rdfs:isDefinedBy .
rdfs:label a owl:AnnotationProperty ;
owl:sameAs rdfs:label .
rdfs:seeAlso a owl:AnnotationProperty ;
owl:sameAs rdfs:seeAlso .
rdfs:subClassOf owl:sameAs rdfs:subClassOf .
xsd:NCName a rdfs:Datatype ;
owl:sameAs xsd:NCName .
xsd:NMTOKEN a rdfs:Datatype ;
owl:sameAs xsd:NMTOKEN .
xsd:Name a rdfs:Datatype ;
owl:sameAs xsd:Name .
xsd:anyURI a rdfs:Datatype ;
owl:sameAs xsd:anyURI .
xsd:base64Binary a rdfs:Datatype ;
owl:sameAs xsd:base64Binary .
xsd:boolean a rdfs:Datatype ;
owl:sameAs xsd:boolean .
xsd:byte a rdfs:Datatype ;
owl:sameAs xsd:byte .
xsd:date a rdfs:Datatype ;
owl:sameAs xsd:date .
xsd:dateTime a rdfs:Datatype ;
owl:sameAs xsd:dateTime .
xsd:dateTimeStamp a rdfs:Datatype ;
owl:sameAs xsd:dateTimeStamp .
xsd:decimal a rdfs:Datatype ;
owl:sameAs xsd:decimal .
xsd:double a rdfs:Datatype ;
owl:sameAs xsd:double .
xsd:float a rdfs:Datatype ;
owl:sameAs xsd:float .
xsd:hexBinary a rdfs:Datatype ;
owl:sameAs xsd:hexBinary .
xsd:int a rdfs:Datatype ;
owl:sameAs xsd:int .
xsd:integer a rdfs:Datatype ;
owl:sameAs xsd:integer .
xsd:language a rdfs:Datatype ;
owl:sameAs xsd:language .
xsd:long a rdfs:Datatype ;
owl:sameAs xsd:long .
xsd:negativeInteger a rdfs:Datatype ;
owl:sameAs xsd:negativeInteger .
xsd:nonNegativeInteger a rdfs:Datatype ;
owl:sameAs xsd:nonNegativeInteger .
xsd:nonPositiveInteger a rdfs:Datatype ;
owl:sameAs xsd:nonPositiveInteger .
xsd:normalizedString a rdfs:Datatype ;
owl:sameAs xsd:normalizedString .
xsd:positiveInteger a rdfs:Datatype ;
owl:sameAs xsd:positiveInteger .
xsd:short a rdfs:Datatype ;
owl:sameAs xsd:short .
xsd:string a rdfs:Datatype ;
owl:sameAs xsd:string .
xsd:time a rdfs:Datatype ;
owl:sameAs xsd:time .
xsd:token a rdfs:Datatype ;
owl:sameAs xsd:token .
xsd:unsignedByte a rdfs:Datatype ;
owl:sameAs xsd:unsignedByte .
xsd:unsignedInt a rdfs:Datatype ;
owl:sameAs xsd:unsignedInt .
xsd:unsignedLong a rdfs:Datatype ;
owl:sameAs xsd:unsignedLong .
xsd:unsignedShort a rdfs:Datatype ;
owl:sameAs xsd:unsignedShort .
owl:backwardCompatibleWith a owl:AnnotationProperty ;
owl:sameAs owl:backwardCompatibleWith .
owl:deprecated a owl:AnnotationProperty ;
owl:sameAs owl:deprecated .
owl:equivalentClass owl:sameAs owl:equivalentClass .
owl:incompatibleWith a owl:AnnotationProperty ;
owl:sameAs owl:incompatibleWith .
owl:priorVersion a owl:AnnotationProperty ;
owl:sameAs owl:priorVersion .
owl:sameAs owl:sameAs owl:sameAs .
owl:versionInfo a owl:AnnotationProperty ;
owl:sameAs owl:versionInfo .
:Guter_Naehrwert_2 a :Guter_Naehrwert,
:Naehrwert,
owl:Thing ;
owl:sameAs :Guter_Naehrwert_2 .
owl:Nothing a owl:Class ;
rdfs:subClassOf :Guter_Naehrwert,
:Kuh,
:Kuhmilch,
:Milch,
:Naehrwert,
:Nahrungsmittel,
:Schlechter_Naehrwert,
:Tier,
:Ziege,
:Ziegenmilch,
owl:Nothing,
owl:Thing ;
owl:equivalentClass owl:Nothing ;
owl:sameAs owl:Nothing .
:Kuh a owl:Class ;
rdfs:subClassOf :Kuh,
:Tier,
owl:Thing ;
owl:equivalentClass :Kuh ;
owl:sameAs :Kuh .
:Kuhmilch a owl:Class ;
rdfs:subClassOf :Kuhmilch,
:Milch,
:Nahrungsmittel,
owl:Thing ;
owl:equivalentClass :Kuhmilch ;
owl:sameAs :Kuhmilch .
:Schlechter_Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert,
:Schlechter_Naehrwert,
owl:Thing ;
owl:equivalentClass :Schlechter_Naehrwert ;
owl:sameAs :Schlechter_Naehrwert .
:Ziege a owl:Class ;
rdfs:subClassOf :Tier,
:Ziege,
owl:Thing ;
owl:equivalentClass :Ziege ;
owl:sameAs :Ziege .
:Guter_Naehrwert a owl:Class ;
rdfs:subClassOf :Guter_Naehrwert,
:Naehrwert,
owl:Thing ;
owl:equivalentClass :Guter_Naehrwert ;
owl:sameAs :Guter_Naehrwert .
:Tier a owl:Class ;
rdfs:subClassOf :Tier,
owl:Thing ;
owl:equivalentClass :Tier ;
owl:sameAs :Tier .
:Ziegenmilch a owl:Class ;
rdfs:subClassOf :Milch,
:Nahrungsmittel,
:Ziegenmilch,
owl:Thing ;
owl:equivalentClass :Ziegenmilch ;
owl:sameAs :Ziegenmilch .
:Naehrwert a owl:Class ;
rdfs:subClassOf :Naehrwert,
owl:Thing ;
owl:equivalentClass :Naehrwert ;
owl:sameAs :Naehrwert .
:Milch a owl:Class ;
rdfs:subClassOf :Milch,
:Nahrungsmittel,
owl:Thing ;
owl:equivalentClass :Milch ;
owl:sameAs :Milch .
:Nahrungsmittel a owl:Class ;
rdfs:subClassOf :Nahrungsmittel,
owl:Thing ;
owl:equivalentClass :Nahrungsmittel ;
owl:sameAs :Nahrungsmittel .
owl:AnnotationProperty owl:sameAs owl:AnnotationProperty .
owl:Class owl:sameAs owl:Class .
owl:Thing a owl:Class ;
rdfs:subClassOf owl:Thing ;
owl:equivalentClass owl:Thing ;
owl:sameAs owl:Thing .
rdfs:Datatype owl:sameAs rdfs:Datatype .
Glossar#
- rdflib#
https://rdflib.readthedocs.io/en/stable/ | https://pypi.org/project/rdflib/
- owlrl#
https://owl-rl.readthedocs.io/en/latest/owlrl.html | https://pypi.org/project/owlrl/ | https://www.w3.org/TR/owl2-profiles/#OWL_2_RL
- SHACL#
Shapes Constraint Language, https://www.w3.org/TR/shacl/ | RDFLib/pySHACL